研究开发FSsgRNA-Analyzer云平台加速CRISPR/Cas技术筛选的数据处理分析
作者:生物谷
日期:2022-09-09
通过全基因组规模扰动文库的构建与筛选,从宏观基因组层面系统研究基因型与表型的对应关系,是微生物功能基因组学研究的重要方法。相较于单个基因扰动文库的构建与筛选,混合文库的构建与筛选可通过一次实验实现特定条件下对上千个基因的同时筛选,显著提高通量。
近年来,CRISPR/Cas技术凭借着精简高效、可编程、易追踪的优势,已逐渐被应用于全基因组规模混合文库的构建与筛选中。但基于DNA双链断裂(Double strand break,DSB)的CRISPR/Cas基因编辑方法由于受DSB的毒性影响,需要添加外源DNA模板且对同源重组效率要求较高,在微生物基因组的文库构建中具有一定的局限性,特别是在同源重组效率普遍偏低的原核生物中更是难以推广。
而基于CRISPR干扰(CRISPR interference,CRISPRi)的文库构建技术则受干扰效率影响较大,作用于多顺反子操纵子时难以定位靶基因。
中国科学院天津工业生物技术研究所研究员王猛带领的高通量编辑与筛选平台实验室与生物设计中心平台实验室合作,结合胞嘧啶碱基编辑技术以及靶向全基因组的sgRNA混合文库,以在基因编码区提前引入终止密码子的方式,开发了全基因组规模单基因失活文库的构建方法,并用于功能基因的筛选。
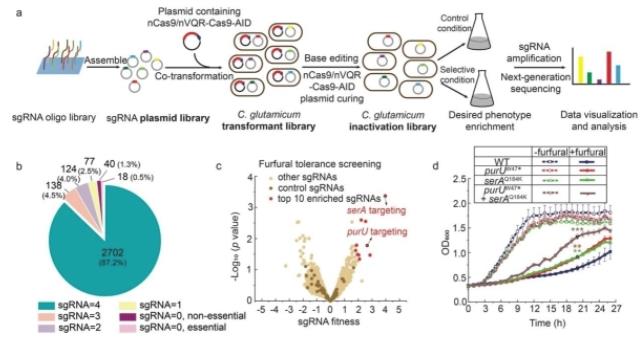
该方法不产生DSB、无需添加外源DNA模板、不依赖宿主的同源重组效率,同时具有操作简单、宿主普适性强、易于推广等优势。
研究人员以重要的工业生产菌株谷氨酸棒杆菌作为示例,设计了约12000条sgRNA,并构建了覆盖度为98.1%的全基因组失活文库,将文库在不同的筛选压力下进行筛选分析。
不同于哺乳动物细胞以及酵母中已报道的数据处理方法,研究人员建立了一套新的sgRNA分析流程,通过对有/无筛选压力下的样品进行二代测序分析,根据sgRNA丰度变化,发掘与筛选性状相关的功能基因。利用上述策略,该研究成功筛选到与5-氟尿嘧啶、5-氟乳清酸抗性,氧化压力耐受、糠醛耐受相关的基因。其中purU以及serA基因首次被鉴定为与糠醛耐受相关。
另外,研究人员开发了FSsgRNA-Analyzer云平台,可加速基于CRISPR/Cas技术筛选的数据处理分析。该研究建立了一种通用性强的基因组文库混合筛选策略,可加快谷氨酸棒杆菌的基础和应用研究,同时为其他原核生物提供了技术参考。


